DNA序列(DNA Consensus String)
输入m个长度为n的DNA序列,求一个DNA序列,到所有序列的总的Hamming距离尽量小,两个等长字符串的Hamming距离等于字符不同的位置个数,例如,ACGT和GCGA的Hamming距离为2(左数第1,4个字符不同)。
输入整数m和n(4<=m<=50,4<=n<=1000),以及m个长度为m的DNA序列(只包含字母A,C,G,T),输出到m个序列的Hamming距离和最小的DNA序列和对应的距离。如有多解,要求为字典序最小的解。例如,对于下面5个DNA序列,最优解为TAAGCTAC。
TATGATAC
TAAGCTAC
AAAGATCC
TGAGATAC
TAAGATGT
#include<stdio.h>#include<string.h>int a[55][55]; //防止程序异常退出int main(){int n,m;char s[55][1005];memset(a,0,sizeof(a));scanf("%d%d",&n,&m);for(int i = 1;i <= n;i++){scanf("%s",s[i]);}//计算Hamming值,for(int i = 1;i <= n;i++){for(int j = 1;j <= n;j++){if(j < i){a[i][j] = a[j][i];}else if(j > i){for(int k = 0;s[i][k] != '\0';k++){if(s[i][k] != s[j][k]){a[i][j]++;}}}}}//统计Hammingfor(int i = 1;i <= n;i++){for(int j = 1;j <= n;j++){a[i][n+1] += a[i][j];}}//找到Hamming最小值的那个DNA序列int min = 1;for(int i = 2;i <= n;i++){if(a[i][n+1] < a[min][n+1])//值小则替换更新{min = i;}else if(a[i][n+1] == a[min][n+1])//值相等则取字典序较小的{if(strcmp(s[i],s[min]) < 0){min = i;}}}printf("%s\n",s[min]);return 0;}


























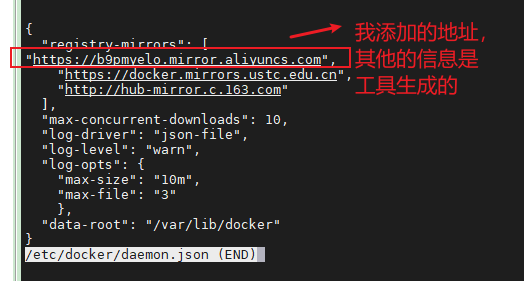
![微信小程序[电商]-撒花完结~ 微信小程序[电商]-撒花完结~](https://image.dandelioncloud.cn/images/20221123/2d6fc62ca0ec49928e1eafdc44c61919.png)




还没有评论,来说两句吧...